🧤 훈련 및 테스트 데이터 분류하기
🧤 훈련, 검증, 테스트, 데이터 분류시 주로 사용되는 변수명
- 정의된 변수 이름은 없음
- 훈련데이터 : 훈련(fit)에 사용되는 데이터
: (훈련 독립변수) train_input, train_x, X_train
: (훈련 종속변수) train_target, train_y, y_train - 검증데이터 : 훈련 정확도(score)에 사용되는 데이터
: (검증 독립변수) val_input, val_x, X_val
: (검증 종속변수) val_target, val_y, y_val - 테스트데이터 : 예측(predict)에 사용되는 데이터
: (테스트 독립변수) test_input, test_x, X_test
: (테스트 종속변수) test_target, test_y, y_test
🧤 데이터 분류 순서
- 훈련과 테스트를 비율로 먼저 나누기
→ 훈련과 테스트 비율 : 주로 7 : 3을 사용, 또는 7.5 : 2.5 또는 8 : 2 - 훈련과 검증 데이터를 나누기
→ 훈련과 검증 비율 : 주로 4 : 2 또는 6 : 2를 사용 - 가장 많이 사용되는 훈련
→ 검증 : 테스트 비율 대략 => 6 : 2 : 2
사용할 데이터 정의하기
fish_length = [25.4, 26.3, 26.5, 29.0, 29.0, 29.7, 29.7, 30.0, 30.0, 30.7, 31.0, 31.0,
31.5, 32.0, 32.0, 32.0, 33.0, 33.0, 33.5, 33.5, 34.0, 34.0, 34.5, 35.0,
35.0, 35.0, 35.0, 36.0, 36.0, 37.0, 38.5, 38.5, 39.5, 41.0, 41.0, 9.8,
10.5, 10.6, 11.0, 11.2, 11.3, 11.8, 11.8, 12.0, 12.2, 12.4, 13.0, 14.3, 15.0]
fish_weight = [242.0, 290.0, 340.0, 363.0, 430.0, 450.0, 500.0, 390.0, 450.0, 500.0, 475.0, 500.0,
500.0, 340.0, 600.0, 600.0, 700.0, 700.0, 610.0, 650.0, 575.0, 685.0, 620.0, 680.0,
700.0, 725.0, 720.0, 714.0, 850.0, 1000.0, 920.0, 955.0, 925.0, 975.0, 950.0, 6.7,
7.5, 7.0, 9.7, 9.8, 8.7, 10.0, 9.9, 9.8, 12.2, 13.4, 12.2, 19.7, 19.9]
🧤 훈련에 사용할 2차원 데이터 형태로 만들기
- 독립변수 정의하기
fish_data = [[l, w]for l, w in zip(fish_length, fish_weight)]
print(fish_data)
len(fish_data)
- 종속변수 정의하기
- 도미 : 빙어 = 1 : 0 -> 찾고자 하는 값을 1로 설정
- 이진분류에서는 찾고자 하는 값을 1로 정의하는 것이 일반적 개념
- 다만, 어떤 값을 사용하여도 무관
fish_target = [1]*35 + [0]*14
print(fish_target)
len(fish_target)
훈련 및 테스트 데이터로 분류하기
🧤 훈련데이터(train)
# - 훈련 독립변수
train_input = fish_data[ : 35]
# - 훈련 종속변수
train_target = fish_target[ : 35]
print(len(train_input), len(train_target))
🧤 테스트 데이터(test)
# - 훈련 독립변수
test_input = fish_data[35 : ]
# - 훈련 종속변수
test_target = fish_target[35 : ]
print(len(test_input), len(test_target))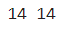
모델 생성하기
from sklearn.neighbors import KNeighborsClassifier
🧤모델(클래스) 생성
- 이웃의 갯수는 기본값 사용
kn = KNeighborsClassifier()
kn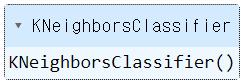
🧤 모델 훈련 시키기
- 훈련데이터 적용
kn.fit(train_input, train_target)
🧤 훈련 정확도 확인하기
# - 훈련 데이터 사용
train_score = kn.score(train_input, train_target)
### 검증하기
# - 테스트 데이터 사용
test_score = kn.score(test_input, test_target)
train_score, test_score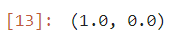
해석
- 훈련 정확도가 1 이기 때문에 과대적합이 발생하였으며, 검증 정확도가 0으로 나타났음
- 따라서, 이 훈련 모델은 튜닝을 통해 성능 향상을 시켜야 할 필요성이 있음
원인분석
- 데이터 분류시 : 35개의 도미값으로만 훈련을 시켰기 때문에 발생한 문제
- 즉, 검증데이터가 0이 나왔다는 것은, 또는 매우 낮은 정확도가 나온 경우 데이터에 편향이 발생하였을 가능성이 있다고 의심해 본다.
- 샘플링 편향 : 특정 데이터에 집중되어 데이터가 구성되어 훈련이 이루어진 경우 발생하는 현상
- 샘플링 편향 해소방법 : 훈련 / 검증 / 테스트 데이터 구성시에 잘 섞어야 한다. (셔플)
샘플링 편향 해소하기(셔플)
🧤 numpy의 셔플링 함수 사용
import numpy as np
🧤 넘파이 배열 형태로 변형하기
input_arr = np.array(fish_data)
tayget_arr = np.array(fish_target)
input_arr
🧤 데이터 갯수 확인하기
- shape : 차원을 확인하는 넘파이 속성(행의 갯수, 열의 갯)
input_arr.shape, tayget_arr.shape
🧤 랜덤하게 섞기
- 랜덤 규칙 지정하기
- random.seed(42) : 데이터를 랜덤하게 생성할 때 규칙성을 띄도록 정의, 숫자 값은 의미없는 값으로 규칙을 의미함
np.random.seed(42)
- index 값 정의하기
index = np.arange(49)
index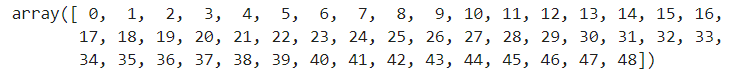
- index 값 셔플로 섞기
np.random.shuffle(index)
index
🧤 훈련 및 테스트 데이터 분류하기
train_input = input_arr[index[ : 35]]
train_target = tayget_arr[index[ : 35]]
test_input = input_arr[index[35 : ]]
test_target = tayget_arr[index[35 : ]]
print(train_input.shape, train_target.shape)
print(test_input.shape, test_target.shape)
🧤 산점도 그리기
### 라이브러리
import matplotlib.pyplot as plt
plt.scatter (train_input[:, 0], train_input[:, 1], c="red", label="train")
plt.scatter(test_input[:, 0], test_input[:, 1], c="blue", label="test")
plt.xlabel("length")
plt.ylabel("weight")
plt.legend()
plt.show()
🧤 모델 생성 및 훈련
### 모델 생성하기
kn = KNeighborsClassifier()
### 훈련시키기
kn.fit(train_input, train_target)
### 훈련 정확도 확인하기
train_score = kn.score(train_input, train_target)
### 테스트 정확도 확인하기
test_score = kn.score(test_input, test_target)
train_score, test_score
🧤 예측하기
test_pred = kn.predict(test_input)
print(f"predict : {test_pred}")
print(f"실제값 : {test_target}")
🧤 하이퍼파라미터 튜닝
### 1보다 작은 가장 좋은 정확도일 때의 이웃의 갯수 찾기
### 모델(클래스 생성)
kn = KNeighborsClassifier()
### 훈련시키기
kn.fit(train_input, train_target)
# - 반복문 사용 : 범위는 3 ~ 전체 데이터 갯수
### 정확도가 가장 높을 떄의 이웃의 갯수를 담을 변수
nCnt = 0
###정확도가 가장 높을때의 값을 담을 변수
nScore = 0
for n in range(3, len(train_input), 2):
kn.n_neighbors = n
score = kn.score(train_input, train_target)
print(f"{n} / {score}")
### 1보다 작은 정확도인 경우
if score < 1 :
### nScore의 값이 score보다 작은 경우 담기
if nScore < score :
nScore = score
nCnt = n
print(f"nCnt = {nCnt} / nScore = {nScore}")
🧤 데이터 섞으면서 분류하기
- 2차원 데이터 생성하기
fish_data = np.column_stack((fish_length,fish_weight))
fish_data
- 1차원 데이터 생성하기
fish_target = np.concatenate((np.ones(35), np.zeros(14)))
fish_target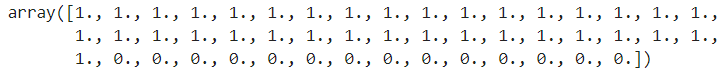
🧤 train_test_split
- 머신러닝, 딥러닝에서 사용하는 데이터 분류기 함수
- 랜덤하게 섞으면서 두개(훈련 : 테스트)의 데이터로 분류함
- 첫번째 값 : 독립변수
- 두번째 값 : 종속변수
- test_size = 0.3 : 분류 기준(훈련 : 테스트 = 7 : 3)
- random_state : 랜덤 규칙
- stratify=fish_target : 종속변수의 범주 비율을 훈련과 테스트의 비율대비 편향 없이 조정시킴
### 라이브러리
from sklearn.model_selection import train_test_split
train_input,test_input, train_target, test_target = train_test_split(fish_data, fish_target, test_size=0.3, random_state=42)
print(f"{train_input.shape}, {train_target.shape} / {test_input.shape}, {test_target.shape}")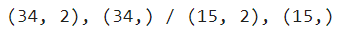
첫번째 결과값 : 훈련 독립변수
두번째 결과값 : 테스트 독립변수
세번째 결과값 : 훈련 종속변수
네번째 결과값 : 테스트 종속변수
🧤 모델 생성 및 훈련
### 모델(클래스 생성하기)
kn = KNeighborsClassifier(n_neighbors=5)
### 모델 훈련시키기
kn.fit(train_input, train_target)
### 훈련 정확도 확인하기
train_score = kn.score(train_input, train_target)
### 검증 정확도 확인하기
test_score = kn.score(test_input, test_target)
train_score, test_score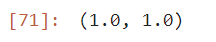
정확도 해석
- 과대적합 : 훈련 > 검증 또는 훈련이 1인 경우
- 과소적합 : 훈련 < 검증 또는 검증이 1인 경우
- 과소적합이 일어나는 모델은 사용할 수 없음
- 과대적합 중에 훈련 정확도가 1인 경우의 모델은 사용할 수 없음
- 과대적합이 보통 0.1 이상의 차이를 보이면 정확도의 차이가 많이난다고 의심해 볼 수 있음
모델 선정 기준
- 과소적합이 일어나지 않으면서, 훈련 정확도가 1이 아니고 훈련과 검증의 차이가 0.1 이내인 경우
- 선정된 모델을 "일반화 모델"이라고 칭한다.
- 다만, 추가로 선정 기준 중에 평가기준이 있음
- 가장 바람직한 결과는 훈련 > 검증> 테스트
- (훈련 > 검증 < 테스트인 경우도 있음)
🧤 하이퍼파라미터 튜닝
### 1보다 작은 가장 좋은 정확도일 때의 이웃의 갯수 찾기
### 모델(클래스 생성)
kn = KNeighborsClassifier()
### 훈련시키기
kn.fit(train_input, train_target)
# - 반복문 사용 : 범위는 3 ~ 전체 데이터 갯수
### 정확도가 가장 높을 떄의 이웃의 갯수를 담을 변수
nCnt = 0
###정확도가 가장 높을때의 값을 담을 변수
nScore = 0
for n in range(3, len(train_input), 2):
kn.n_neighbors = n
score = kn.score(train_input, train_target)
print(f"{n} / {score}")
### 1보다 작은 정확도인 경우
if score < 1 :
### nScore의 값이 score보다 작은 경우 담기
if nScore < score :
nScore = score
nCnt = n
print(f"nCnt = {nCnt} / nScore = {nScore}")
🧤 임의로 테스트 하기 (for문 돌리지 않기)
kn.predict([[25, 150]])
🧤 산점도 그리기
plt.scatter(train_input[:, 0], train_input[:, 1], c="red", label="bream")
plt.scatter(25, 150, marker='^', c="green", label="pred")
plt.xlabel("length")
plt.ylabel("weight")
plt.legend()
plt.show()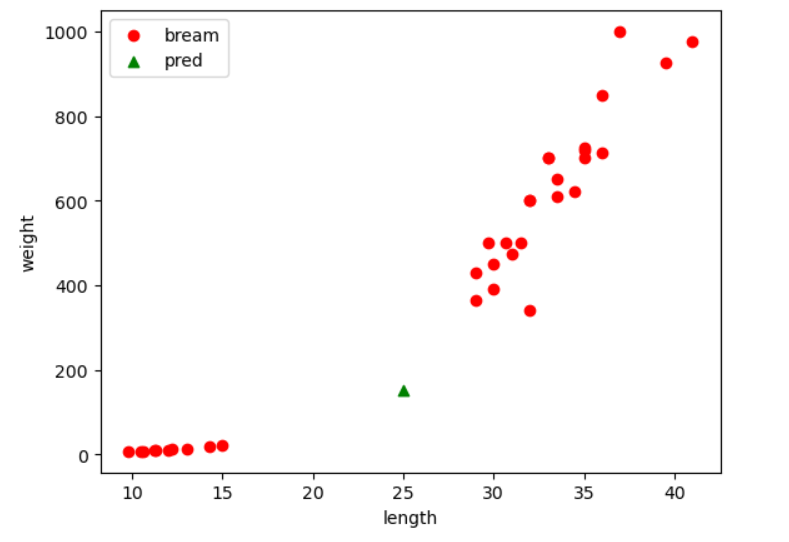
🧤 사용된 이웃 확인하기
- kneighbors(이웃과의 거리, index 위치값)
dist, indexes = kn.kneighbors([[25, 150]])
indexes
- 이웃을 포함하여 산점도 그리기
plt.scatter(train_input[:, 0], train_input[:, 1], c="red", label="bream")
plt.scatter(25, 150, marker='^', c="green", label="pred")
plt.scatter(train_input[indexes, 0], train_input[indexes, 1], c="blue", label="nei")
plt.xlabel("length")
plt.ylabel("weight")
plt.legend()
plt.show()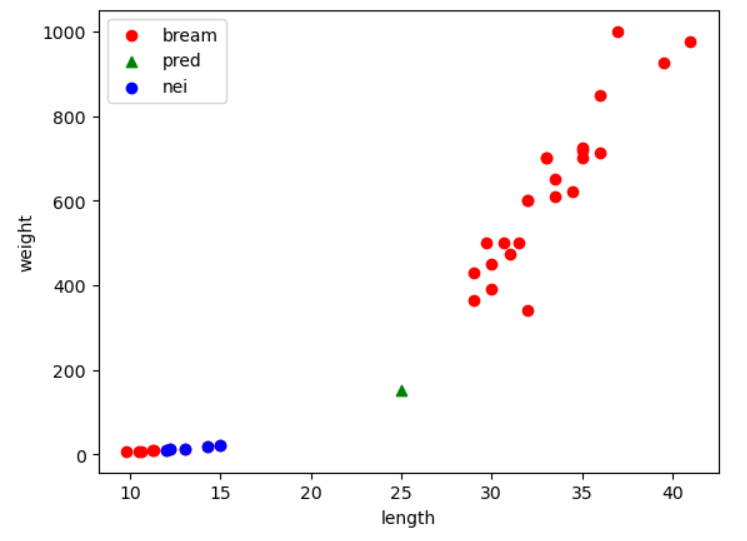
분석
- 예측 결과는 빙어로 확인되었으나, 시각적으로 확인하였을 때는 도미에 더 가까운 것으로 확인 됨
- 실제 이웃을 확인한 결과 빙어쪽 이웃을 모두 사용하고 있음
- 이런 현상이 발생한 원인 : 스케일(x축과 y축의 단위)이 다르기 때문에 나타나는 현상 → "스케일이 다르다" 라고 표현 한다.
- 해소 방법 : 데이터 정규화 전처리를 수행해야 함
🧤 정규화 하기
현재까지 수행 순서
1. 데이터 수집
2. 독립변수 2차원과 종속변수 1차원 데이터로 취합
3. 훈련, 검증, 테스트 데이터로 섞으면서 분리
4. 훈련, 검증, 테스트 데이터 중에 독립변수에 대해서만 정규화 전처리 수행
5. 훈련모델 생성
6. 모델 훈련 시키기
7. 훈련 및 검증 정확도 확인
8. 하이퍼파라미터 튜닝
9. 예측
정규화
🧤 정규화 → 표준점수화 하기
- 표준점수 = (각 데이터 - 데이터 전체 평균) / 데이터 전체 표준편차
- 표준점수 : 각 데이터가 원점(0)에서 표준편차 만큼 얼마나 떨어져 있는지를 나타내는 값
🧤 데이터 전체 평균 구하기
mean = np.mean(train_input, axis=0)
mean
🧤 데이터 전체 표준편차 구하기
std = np.std(train_input, axis=0)
std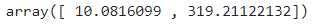
🧤정규화(표준점수) 처리하기
train_scaled = (train_input - mean) / std
train_scaled
🧤 이웃을 포함하여 산점도 그리기
plt.scatter(train_scaled[:, 0], train_scaled[:, 1], c="red", label="bream")
plt.scatter(25, 150, marker='^', c="green", label="pred")
plt.scatter(train_input[indexes, 0], train_input[indexes, 1], c="blue", label="nei")
plt.xlabel("length")
plt.ylabel("weight")
plt.legend()
plt.show()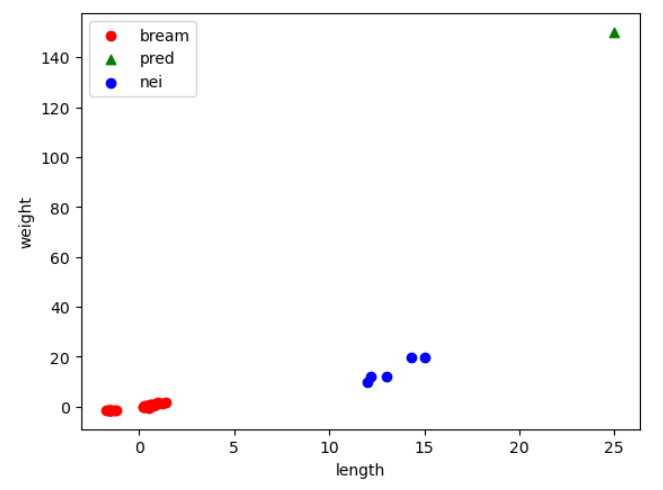
🧤 예측하고자 하는 값들도 모두 정규화 처리해야 함
new = ([25, 150] - mean) / std
new
🧤 이웃을 포함하여 산점도 그리기
plt.scatter(train_scaled[:, 0], train_scaled[:, 1], c="red", label="bream")
plt.scatter(new[0], new[1], marker='^', c="green", label="pred")
plt.scatter(train_scaled[indexes, 0], train_scaled[indexes, 1], c="blue", label="nei")
plt.xlabel("length")
plt.ylabel("weight")
plt.legend()
plt.show()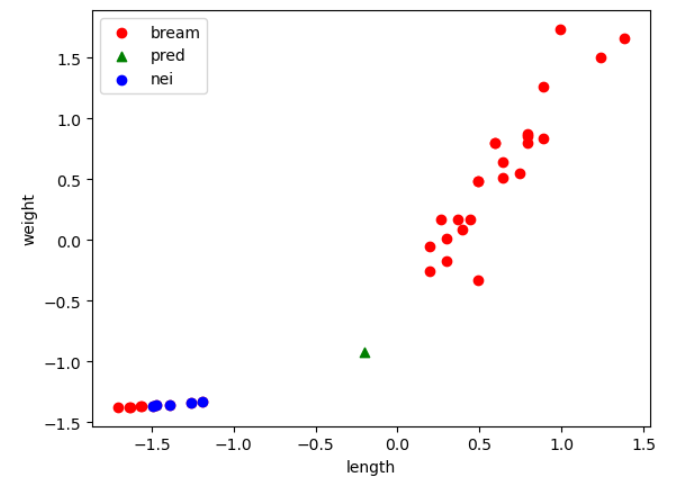
모델 훈련시키기
### 모델(클래스 생성하기)
kn = KNeighborsClassifier()
### 모델 훈련시키기
kn.fit(train_scaled, train_target)
### 훈련 정확도 확인하기
train_score = kn.score(train_scaled, train_target)
train_score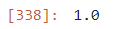
🧤 테스트 데이터로 검증하기
- 검증 또는 테스트 데이터를 스케일링 정규화 처리
- 이때는 훈련에서 사용한 mean과 std 그대로 사용해야 함
test_scaled = (test_input - mean) / std
test_score = kn.score(test_scaled, test_target)
test_score
🧤 예측하기
kn.predict([new])
🧤 예측에 사용된 이웃 확인하고 시각화 하기
dist , indexes = kn.kneighbors([new])
indexes
🧤 이웃을 포함하여 산점도 그리기
plt.scatter(train_scaled[:, 0], train_scaled[:, 1], c="red", label="bream")
plt.scatter(new[0], new[1], marker='^', c="green", label="pred")
plt.scatter(train_scaled[indexes, 0], train_scaled[indexes, 1], c="blue", label="nei")
plt.xlabel("length")
plt.ylabel("weight")
plt.legend()
plt.show()
'Digital Boot > 인공지능' 카테고리의 다른 글
| [인공지능][ML] Machine Learning - 분류모델 / 앙상블모델 / 배깅 / 부스팅 / 랜덤포레스트 (0) | 2023.12.26 |
|---|---|
| [인공지능][ML] Machine Learning - 선형회귀모델 / 다항회귀모델 / 다중회귀모델 (0) | 2023.12.21 |
| [인공지능][ML] Machine Learning - KNN 회귀모델 / 평균절대오차(MAE) (0) | 2023.12.21 |
| [인공지능][ML] Machine Learning - K최근접이웃모델 / KNN (5) | 2023.12.20 |
| [인공지능] Machine Learning - 개요 / 실습 (3) | 2023.12.19 |